Wir evaluieren in den ersten 24 Stunden nach Erscheinen alle makromolekularen Strukturen aus SARS-CoV und SARS-CoV-2, damit Downstream-Verwender die bestmöglichen Daten haben.
Die Skripte zum Herunterladen einzelner Strukturen sind unter https://github.com/thorn-lab/coronavirus_structural_task_force/tree/master/utils/Update_pipeline zu finden.
Vergleich zwischen dem Modell und dem Vorwissen über Protein- und RNA-Strukturen
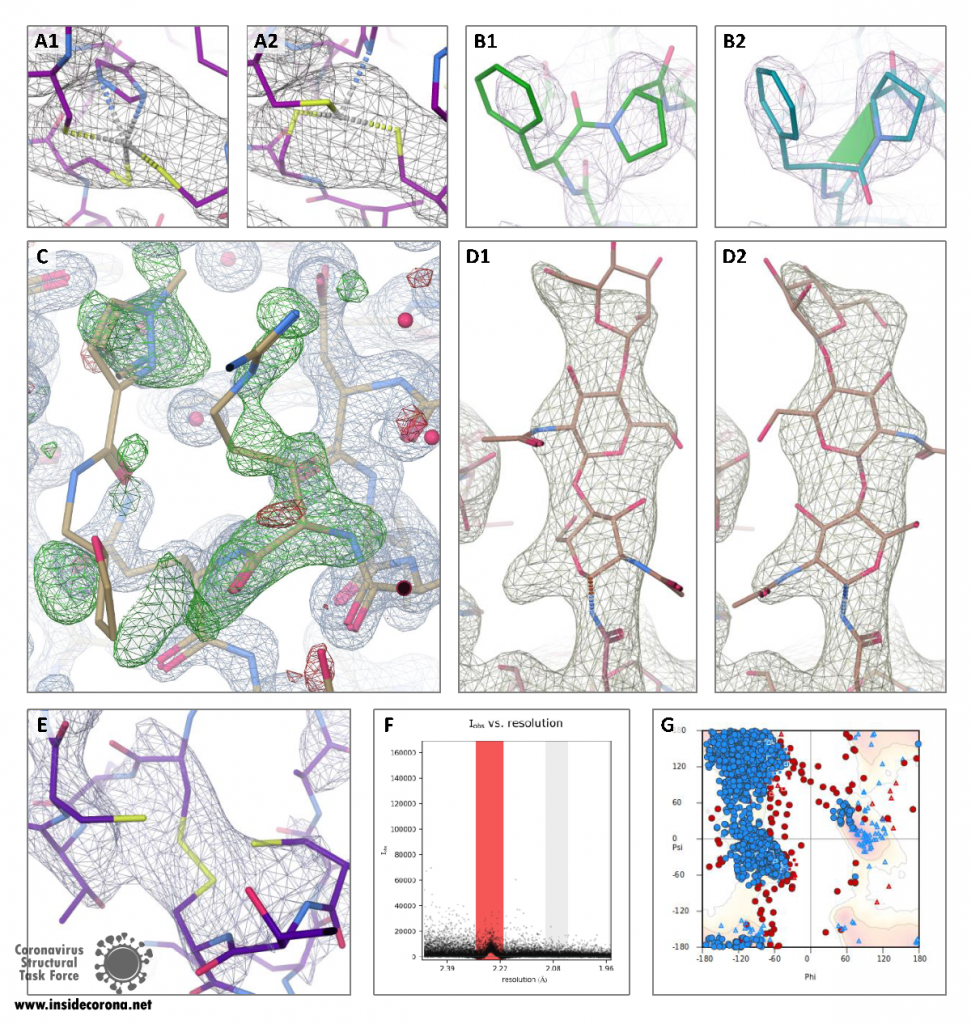
Die Molekulargeometrie ist durch chemische Bindungen und sterische Hinderung beschränkt. Wir verwenden MolProbity, um die Modellqualität unter den Gesichtspunkten unseres chemischen Vorwissens zu bewerten. MolProbity überprüft die Bindungen und daraus resultierende Geometrie, Konformation von Proteinen und RNA sowie sterische Kollisionen. Leider können diese traditionellen Indikatoren für die Modellqualität auch als Restraints im Modellbau verwendet werden und erlauben dann natürlich keine unabhängige Aussage mehr über die Modellqualität. Aus diesem Grund berechnen wir zusätzlich die CaBLAM-Score, die es auch in solchen Fällen gestattet, lokale Fehler bei einer Auflösung von 3-4 Å zu finden. 163 unserer Strukturen haben eine CaBLAM-Score höher als 2%. Die betreffenden Strukturen haben fehlerhafte Hauptketten-Konformationen.
Während der ersten Monate der Pandemie wurde die Kapazität des MolProbity-Webdienstes ausgereizt, weil viele verschiedene Medikamentenentwickler immer dieselben Coronavirus-Strukturen validiert haben. Auch aus diesem Grund haben wir eine maßgeschneiderte MolProbity-Pipeline entwickelt, um diese Ergebnisse für die Corona-Strukturen online verfügbar zu machen.
Darüber hinaus überprüfen wir die Qualität der experimentellen Daten und wie gut das Modell zu diesen Daten passt:
Für Diffraktions-Daten
Kristallstrukturen machen den Großteil unserer Daten aus und werden deswegen am gründlichsten ausgewertet. Die Diffraktion kann z.B. von mehr als einem Kristallgitter stammen (Verzwillingung), durch Eiskristallbeugung verunreinigt sein (Eisringe) oder aufgrund von Strahlenschäden oder suboptimaler Messstrategie unvollständig sein. Diese Probleme können nach der Messung nicht mehr vermieden werden, aber eine entsprechende Prozessierung der Daten kann zu einem besseren Strukturmodell führen. Die Identifikation solcher Probleme aus den prozessierten Messdaten kann schwierig sein; Rohdaten erlauben eine viel vollständigere Analyse des Experiments, werden aber nicht routinemäßig veröffentlicht. - Denn die Datenprozessierung (Integration und Skalierung) ist eine weitere Fehlerquelle; sie erfolgt heutzutage oft automatisch. Die Annahme einer falschen Kristallgittersymmetrie oder die Berechnung von Intensitäten von im Experiment eigentlich verdeckten Reflexen kann zu qualitativ schlechteren oder sogar unlösbaren Strukturen führen. Wenn Rohdaten aber verfügbar sind, können die Daten erneut prozessiert werden, um solche Probleme zu beheben. Wir verwenden phenix.xtriage, um kristallographische Daten auf Verzwillingung, Vollständigkeit und allgemeine Beugungsqualität zu überprüfen. Die Log-Dateien befinden sich im Unterverzeichnis validation/ im Strukturverzeichnis. AUSPEX identifiziert automatisch Eisringe und erstellt Plots, aus denen verschiedene andere Pathologien, wie z.B. eine schlecht angepasste oder fehlende Primärstrahlfänger-Maske schnell erkannt werden können. Die Plots von AUSPEX und die entsprechenden (nicht automatischen) Kommentare befinden sich in validation/auspex.
Ein allgemeiner Anhaltspunkt dafür, wie gut das Atommodell und die Messdaten zusammenpassen, kann durch die Bewertung der R-Faktoren erfolgen. Auch ein Vergleich der hinterlegten R-Faktoren mit den Ergebnissen von PDB-REDO (einschließlich Whatcheck) kann weiterhelfen. Die PDB-REDO und Whatcheck-Ergebnisse befinden sich in validation/pdb-redo unter dem PDB-Eintragsverzeichnis. Obwohl PDB-REDO die Strukturen oft verbessert, müssen sie überprüft werden und sollten nicht allein auf der Basis eines niedrigeren R-Wertes als "richtiger" angesehen werden. Darüber hinaus weist ein hoher R-Wert nicht auf spezifische Fehler hin; diese müssen manuell gefunden werden.
Für Kryo-EM-Daten:
Kryo-EM-Strukturen machen etwa 15% unserer Daten aus. Wie bei kristallographischen Strukturen sind keine Rohdaten in der wwPDB verfügbar, sondern es wird die aus den Einzelpartikelbildern rekonstruierte dreidimensionale Dichte hinterlegt. Sie ermöglicht eine Berechnung der Passung zwischen Modell und Dichte als Fourier-Shell-Korrelation (FSC). FSC wird als Kurve gegen die Auflösung aufgetragen. Für ein gut gepasstes Modell entspricht ein FSC von 0,5 ungefähr der Auflösung der Kryo-EM-Dichte (welche aus der FSC zwischen zwei Dichten, die aus je der Hälfte der Einpartikelbilder generiert wurde, bestimmt wird. Der Cut-off hierbei ist 0,143). Wir verwenden zur Berechnung das CCP-RM Model Validation Task. Diese Pipeline berechnet auch den Cross-Correlation-Koeffizienten (CCC) für den realen Raum, den Mutual Information (MI) und den Segment Manders’ Overlap Coefficient (SMOC) berechnet. Während der MI ein einzelner Wert für die Übereinstimmung zwischen Modell und Dichte ist, bewertet der SMOC-Score die Passung jedes modellierten Restes einzeln und kann so dabei helfen, schlecht passende Regionen im Modell zu finden. Z-Scores weisen auf diese hin.
Zusätzlich verwenden wir Haruspex, ein neuronales Netz zur Annotation von Sekundärstruktur in Rekonstruktionsdichte, um Sekundärstrukturen in der Dichte zu annotieren, die dann mit dem Modell verglichen werden können.
Manuelle Analyse und Verbesserung von Strukturen
Von den Hunderten SARS-CoV-2 Strukturen, die seit Beginn der Pandemie aufgeklärt wurden, können wir nur einen Teil von Hand bewerten. Aus diesem Grund haben wir repräsentative Strukturen von allen Proteinen ausgewählt und sie Atom für Atom evaluiert, und stellen, wenn nötig, ein verbessertes Modell zur Verfügung. Diese Strukturen kann man hier finden.
Zusammenfassung aller Informationen
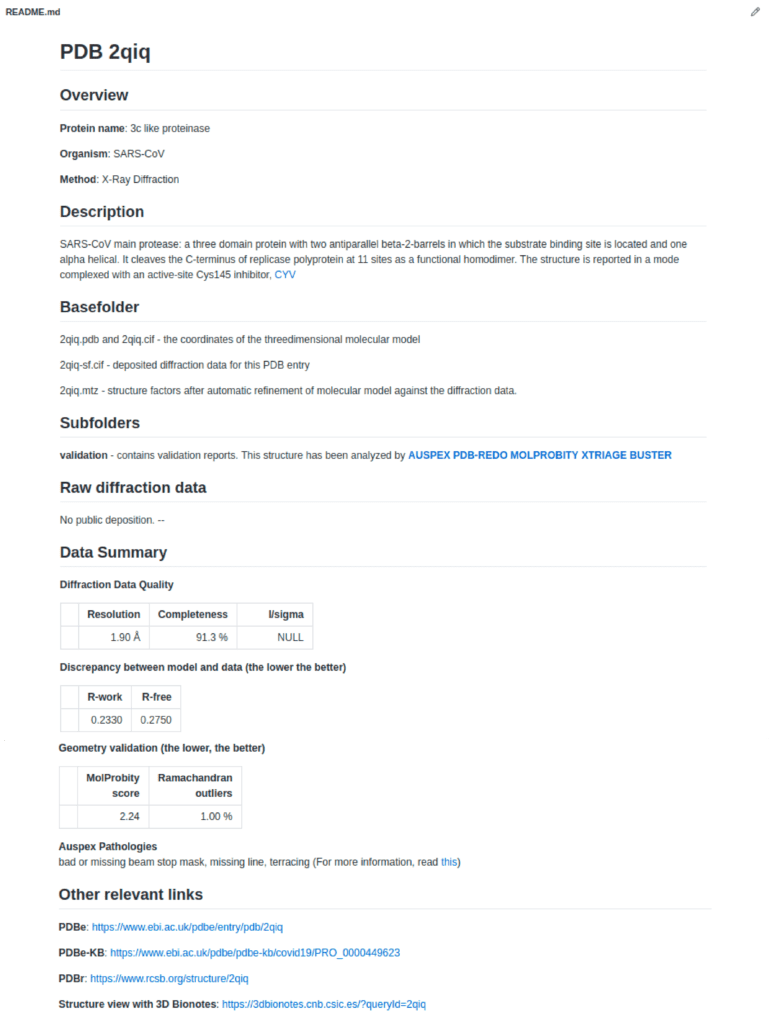
Die wichtigsten Informationen über jede einzelne Struktur sind in der Datei README.md (in jedem PDB-Eintragsverzeichnis) zusammengestellt. Online-Benutzer können die Berichte direkt im Browser anzeigen lassen.
Eine Gesamtbeschreibung wird von unseren Spezialisten für Struktur-/Molekularbiologie auf der Grundlage des Depositionsberichts sowie der relativen Literatur des Struktureintrags erstellt. Die Beschreibung umfasst die allgemeine Funktion des Proteins, den beabsichtigten Zweck der Lösung einer solchen Struktur, die potenziellen Bindungsliganden und Schlussfolgerungen (falls vorhanden), die man aus dem Modell ziehen kann. Hinzu kommt eine kurze Beschreibung gefundener Probleme und der Validierungsergebnisse.
Falls Rohdaten (Diffraktionsbilder für Kristallographie und Mikroskop-aufnahmen für Kryo-EM) verfügbar sind, so werden diese aus der Datenbank verlinkt. Weitere nützliche Links, zum Beispiel zur PDBe Knowledgebase oder zum 3D Bionotes Structural viewer sind auch angegeben.